AI蛋白質(zhì)設(shè)計前沿教程,AAAI'25三大機構(gòu)攜手4小時全面剖析
精準預測和設(shè)計蛋白質(zhì)的序列、結(jié)構(gòu)及模擬其動態(tài)變化,一直是科學界的重大挑戰(zhàn)。
在即將舉行的AAAI 2025會議上,加拿大魁北克省人工智能研究所Mila、美國東北大學和MIT的學者將組織一場主題為“人工智能在蛋白質(zhì)設(shè)計中的應用”的教程。
(文末附教程直通車)
綜觀當下,AI與生命科學深度融合背景下,蛋白質(zhì)研究正經(jīng)歷前所未有的AI驅(qū)動變革。
作為生命活動的核心,蛋白質(zhì)在細胞結(jié)構(gòu)構(gòu)建、物質(zhì)運輸和催化化學反應中扮演著關(guān)鍵角色。如今,AI技術(shù)的介入,以前所未有的速度和力度,重塑了蛋白質(zhì)研究的格局,帶來了無限可能。
這不僅加速了新藥研發(fā)和生物技術(shù)創(chuàng)新,也為解決環(huán)境和工業(yè)領(lǐng)域的挑戰(zhàn)提供了新的工具。
本次教程將全面回顧AI在蛋白質(zhì)預測與設(shè)計領(lǐng)域的最新進展,探討當前的研究成果和未來的發(fā)展方向。
同時,教程將展望AI在蛋白質(zhì)設(shè)計中的未來趨勢,討論可能面臨的挑戰(zhàn)和機遇。

無論是蛋白質(zhì)序列表示學習,還是結(jié)構(gòu)研究,AI都展現(xiàn)出巨大潛力。
本次教程中,主辦方將詳細介紹AI在蛋白質(zhì)序列、結(jié)構(gòu)和功能預測與設(shè)計中的應用方法。通過生成模型進行蛋白質(zhì)設(shè)計,甚至可以創(chuàng)造具有特定功能的新型蛋白質(zhì)。
教程希望參與者具備機器學習的基礎(chǔ)知識,但即使缺乏計算生物學或生物信息學經(jīng)驗也無妨,課程將提供入門介紹,幫助大家了解這一交叉學科領(lǐng)域。
教程定于美東時間2月26日上午8:30至中午12:30在賓夕法尼亞州費城會議中心117號房間舉行。
教程大綱:多維度解鎖蛋白質(zhì)設(shè)計奧秘
本次教程將從多個維度深入解析 AI 蛋白質(zhì)設(shè)計領(lǐng)域的關(guān)鍵問題、前沿方法和研究趨勢,具體內(nèi)容如下:
(一)序章:前沿洞察與基礎(chǔ)知識
本部分將重點介紹AI在蛋白質(zhì)研究領(lǐng)域取得的重大突破。
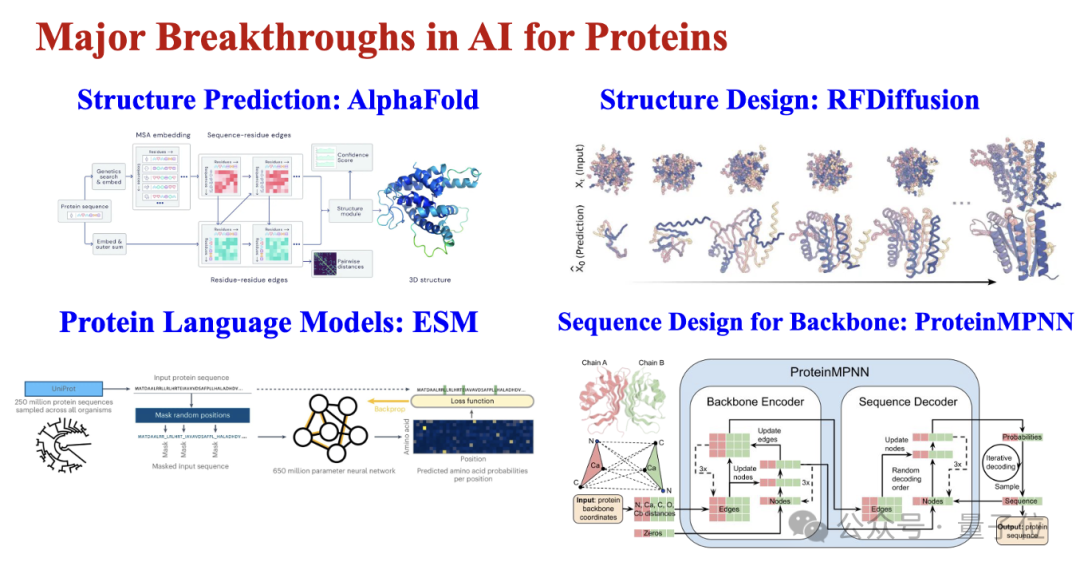
從早期探索到最新成果,AI 在蛋白質(zhì)研究中的每一步進展都意義深遠。
同時,將對蛋白質(zhì)的基本結(jié)構(gòu)、功能及其在生命活動中的重要角色進行科普講解。
最后,討論如何對蛋白質(zhì)數(shù)據(jù)進行學習,為后續(xù)深入研究奠定堅實基礎(chǔ)。
(二)蛋白質(zhì)表示學習:挖掘數(shù)據(jù)的潛在價值
本部分是教程的核心之一,將詳細介紹多種蛋白質(zhì)表示學習的方法。

在序列表示學習中,將深入探討自回歸語言模型、掩碼語言模型和擴散語言模型,這些模型以不同方式對蛋白質(zhì)序列進行編碼,提取關(guān)鍵信息。
結(jié)構(gòu)表示學習方面,幾何深度學習為研究蛋白質(zhì)的幾何結(jié)構(gòu)提供了新視角;GVP、GearNet等蛋白質(zhì)結(jié)構(gòu)編碼器,以及多種結(jié)構(gòu)預訓練算法,從不同角度對蛋白質(zhì)結(jié)構(gòu)進行學習和優(yōu)化。
多模態(tài)表示學習結(jié)合了序列、結(jié)構(gòu)、功能和文本等多種信息,模型如ESM-GearNet、SaProt、DPLM-2、ESM3、ProtST等,展示了多模態(tài)融合在蛋白質(zhì)研究中的強大能力。
最后,將介紹這些方法在蛋白質(zhì)理解任務、蛋白質(zhì)適應性預測和抗體親和力優(yōu)化等方面的應用。
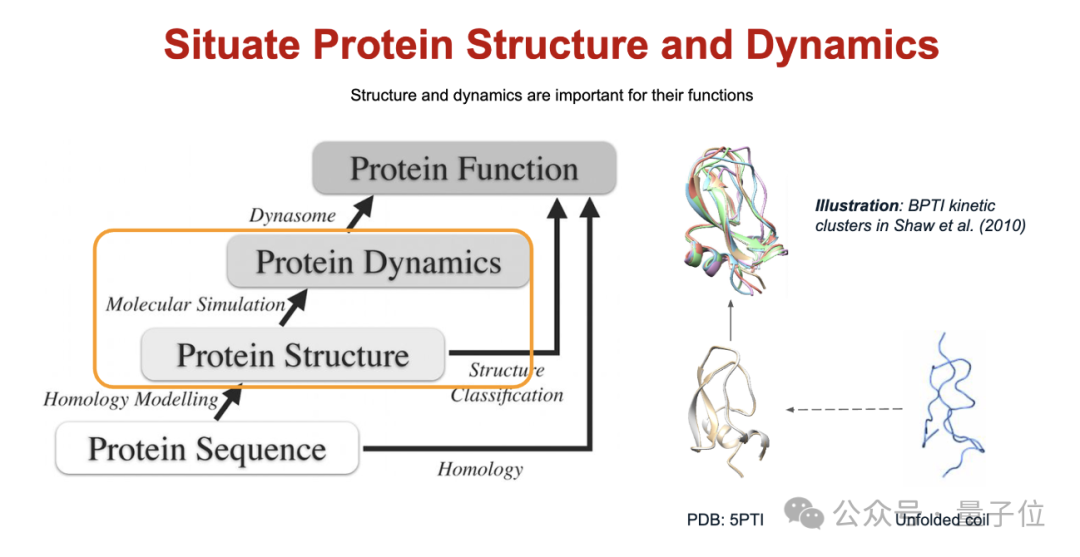
(三)蛋白質(zhì)結(jié)構(gòu)和動力學預測,探索分子動態(tài)奧秘
蛋白質(zhì)結(jié)構(gòu)和動態(tài)預測是研究中的核心問題之一。
本部分將介紹單鏈折疊(如 AlphaFold2、ESMFold)、側(cè)鏈預測(如 AttnPacker、DiffPack)和復合物預測(如 AlphaFold-Multimer、AlphaFold3)等前沿方法。
在蛋白質(zhì)構(gòu)象采樣方面,與會人員將探討玻爾茲曼生成器、基于粗粒度的方法、基于剛性框架的方法以及蛋白質(zhì)結(jié)構(gòu)語言模型。
在分子動力學(MD)軌跡模擬中,將介紹神經(jīng)模擬器、條件轉(zhuǎn)移算子和軌跡生成器等前沿方法。
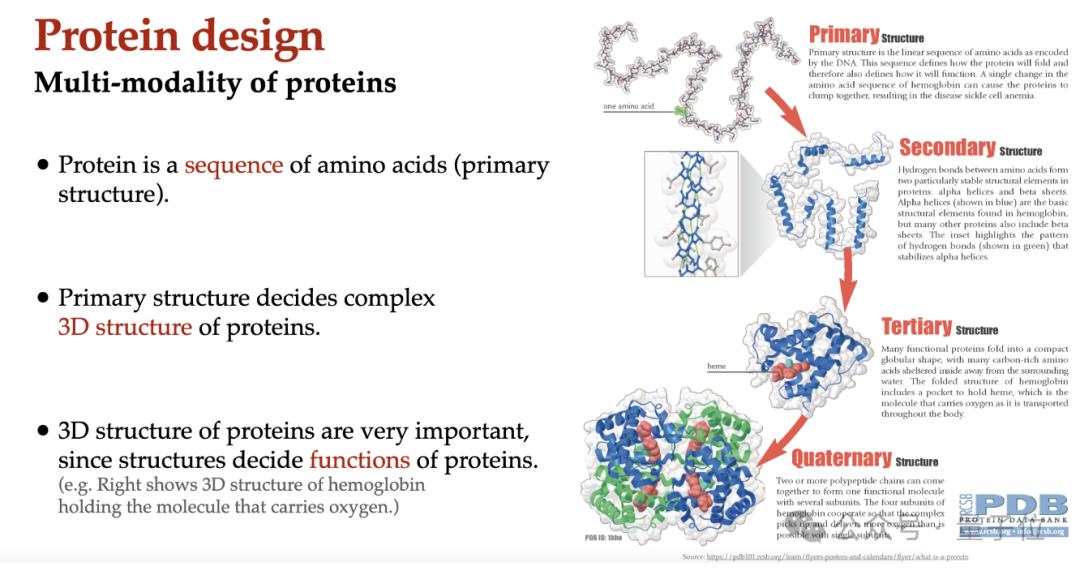
(四) 蛋白質(zhì)設(shè)計:開啟新型蛋白質(zhì)創(chuàng)造之門
在序列設(shè)計方面,主辦方將介紹無條件序列生成(如ProGen)和逆折疊(如ESM-IF、ProteinMPNN)的方法。
結(jié)構(gòu)設(shè)計中,F(xiàn)rameDiff、FrameFlow、Genie2、Chroma、RFDiffusion、FoldFlow、FoldFlow-2等模型將是討論的重點。
序列-結(jié)構(gòu)協(xié)同設(shè)計部分,ProtSeed、ProteinGenerator、MultiFlow、Protpardelle、DPLM-2 等模型將展示如何同時優(yōu)化蛋白質(zhì)的序列和結(jié)構(gòu)。
抗體設(shè)計將聚焦于RefineGNN、AbX等方法。
(五)總結(jié)與展望:共繪蛋白質(zhì)設(shè)計的未來藍圖
這一部分將系統(tǒng)回顧整個教程的核心內(nèi)容,梳理人工智能在蛋白質(zhì)設(shè)計領(lǐng)域的最新進展與取得的成果。
此外,主辦方還將對該領(lǐng)域未來可能的發(fā)展趨勢進行展望,深入探討前沿研究中可能遇到的挑戰(zhàn)及潛在的機遇。
最后,教程特別設(shè)置了互動問答環(huán)節(jié),以進一步增進交流、啟發(fā)思考,共同探索蛋白質(zhì)設(shè)計研究的新方向。
教程背后組織團隊
本次教程由加拿大Mila教授唐建,和美國東北大學教授金汶功團隊聯(lián)合組織。
唐建博士是加拿大魁北克省人工智能研究中心Mila副教授,該中心由圖靈獎獲得者、“AI之父” Yoshua Bengio 創(chuàng)立。
他還是加拿大CIFAR AI講席教授、北京百奧幾何公司創(chuàng)始人兼首席執(zhí)行官。
此外,唐建是圖表示學習領(lǐng)域的知名學者,在深度生成模型、圖機器學習及其藥物發(fā)現(xiàn)應用方面成果顯著。
他曾發(fā)表圖表示學習領(lǐng)域的經(jīng)典論文LINE(單篇引用次數(shù)超過6000次),并將這些技術(shù)開創(chuàng)性地應用于藥物發(fā)現(xiàn)領(lǐng)域,發(fā)表了一系列極具影響力的代表作,包括最早用于分子圖的預訓練算法InfoGraph、第一個用于分子三維結(jié)構(gòu)生成的擴散生成模型ConfGF和GeoDiff,以及最早基于蛋白質(zhì)三維結(jié)構(gòu)的預訓練模型之一GearNet。
他還與英偉達、Intel、IBM 等機構(gòu)共同開發(fā)了業(yè)內(nèi)首個專門針對藥物與蛋白質(zhì)的開源機器學習平臺TorchDrug和TorchProtein。相關(guān)研究發(fā)表于Nature、PNAS、Nature Communications、Nature Machine Intelligence、NeurIPS、ICML、ICLR等頂級會議與期刊。
同時,唐建還擔任 NeurIPS 和 ICML 的領(lǐng)域主席,以及機器學習領(lǐng)域著名期刊 Journal of Machine Learning Research (JMLR) 的執(zhí)行編輯。
金汶功博士是美國東北大學Khoury計算機科學學院助理教授,同時擔任Broad Institute Eric and Wendy Schmidt 中心的訪問研究科學家。
他于MIT CSAIL獲得博士學位,導師為Regina Barzilay和Tommi Jaakkola。
在AI藥物發(fā)現(xiàn)領(lǐng)域的算法創(chuàng)新方面,金汶功團隊等變神經(jīng)網(wǎng)絡(luò)、擴散模型等多個方向有所建樹,開發(fā)出RefineGNN、Mol2Image等模型與算法,成果發(fā)表于NeurIPS、ICLR等頂會。
在藥物發(fā)現(xiàn)方面,團隊成功發(fā)現(xiàn)了新型抗生素,成果發(fā)表在Cell和 Nature等。
在化學工程領(lǐng)域,團隊還助力實現(xiàn)自動化化學合成實驗室,其開發(fā)的化學反應結(jié)果預測算法精度達到化學家級別,相關(guān)成果發(fā)表于NeurIPS和Chemical Science等期刊。