初探AI拼圖模型預測蛋白質復合物結構
分子表示學習在 AI 輔助藥物發現研究中起著至關重要的作用。在傳統藥物研發中,常用的分子對接模型需要進行大量的構型采樣與優化,并篩選出較為穩定的結構。這類策略效率較低,難以應用于高通量的蛋白質對接任務。本文介紹的基于分子表面黎曼流形的深度學習表示方法 (Harmonic Molecular Representation, HMR) 實現了更準確、高效的蛋白質對接模型開發。HMR 用二維黎曼流形建模分子表面,結合調合分析技術與神經網絡實現流形上幾何、化學信號的多尺度傳播和兩個蛋白質表面之間的匹配度比較,進而利用「蛋白質拼圖」的邏輯實現蛋白質分子剛性對接 (rigid protein docking) 。實驗表明,基于 HMR 的分子對接模型比當前深度學習 SOTA [1] 有更高的準確性,并且較傳統分子對接方法提速 100 倍以上。相關論文已被收錄于 ICLR 2023。
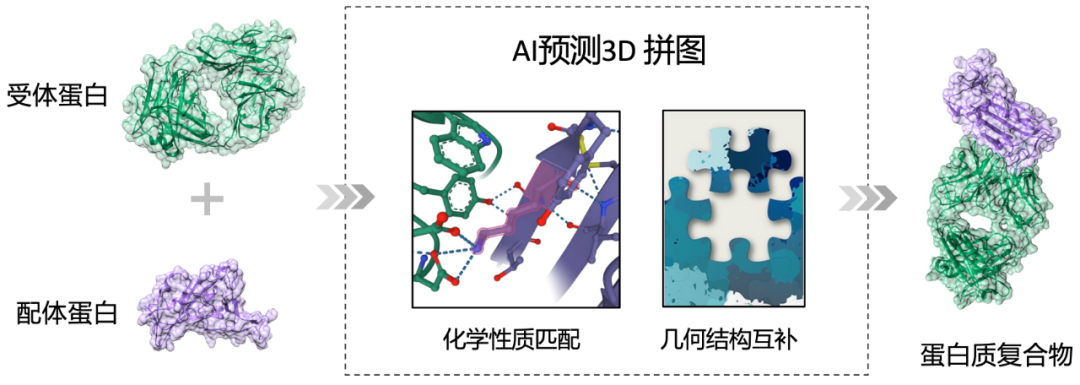
蛋白質之間的相互作用是實現其生物活性的重要基礎,例如人體可以產生抗體蛋白(上圖綠色部分)與入侵的病菌(紫色部分)結合從而抑制疾病。生物制藥研究通過分析生物分子之間相互作用的物理、化學機制,進一步設計出可以與一些特定靶點相結合的新型藥物分子(如研發新冠抗體)。在微觀尺度下,蛋白質之間的相互結合主要由分子間作用力決定,例如氫鍵、靜電力、疏水作用等。
在傳統藥物研發中,分子對接技術 (molecular docking) 通過物理方法建模這些微觀作用來模擬計算真實生物體內兩個分子相互作用的穩定結構。這些傳統的分子對接模型需要進行大量的構型采樣及優化,并篩選出其中較為穩定的結構作為預測結果。這種基于采樣和篩選的策略導致傳統方法效率較低,難以應用于高通量的蛋白質對接任務(例如從 1 萬個蛋白中篩選出最有可能與病菌表面結合的 10 個來進行濕實驗測試)。研發準確且高效的分子對接模型可以幫助快速篩選出適合進行濕實驗測試的蛋白質分子,從而提高新藥研發的效率。
為了開發更加準確且高效的蛋白質對接模型,字節跳動 ByteDance Research 團隊設計了一套基于分子表面的幾何深度學習方案。該方案的核心思想是訓練 AI 模型從拼圖的角度去理解蛋白質之間的相互作用,并按照拼圖的思路實現蛋白質復合物構型預測。

- 論文地址:https://openreview.net/pdf?id=ySCL-NG_I3
- 代碼地址:https://github.com/GeomMolDesign/HMR
具體來說,如果兩個蛋白質可以結合,那么其結合區域必須同時滿足化學性質匹配和幾何結構互補這兩個條件(下圖)。因此直觀上可以將其類比為一個拼圖問題:只要能夠在蛋白質表面分別找到兩塊形狀和紋理都能匹配的「拼圖」,那么就可以將這兩個蛋白質拼在一起形成穩定的蛋白質復合物。
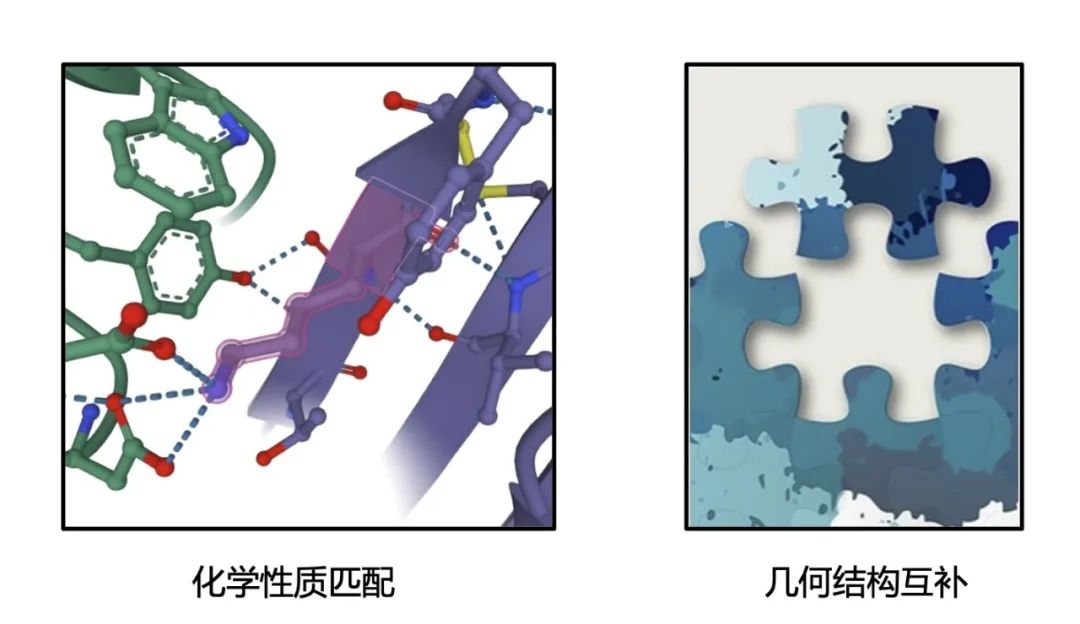
圖:能夠結合的蛋白質分子必須同時滿足化學性質匹配和幾何結構互補這兩個條件,類似于拼圖問題。
基于上述假設,團隊提出了一種基于分子表面黎曼流形的深度學習表示方法 (Harmonic Molecular Representation, HMR) :用二維黎曼流形建模分子表面,結合調合分析技術與神經網絡實現流形上幾何、化學信號的多尺度傳播和兩個蛋白質表面之間的匹配度比較,進而利用「蛋白質拼圖」的邏輯實現蛋白質分子剛性對接 (rigid protein docking) 。直觀上,神經網絡模型需要從大量蛋白質復合物的結構(訓練集)中學習這類拼圖的幾何、化學規律,從而可以去預測沒有見過的(測試集)蛋白質復合物結構。
實驗表明,基于 HMR 的分子對接模型比當前深度學習 SOTA [1] 有更高的準確性,并且較傳統分子對接方法提速 100 倍以上。接下來的 3 個章節將分別介紹黎曼流形分子表示、分子表面幾何深度學習和蛋白質拼圖模型的主要思想和技術概要。
§1 基于黎曼流形的分子表示
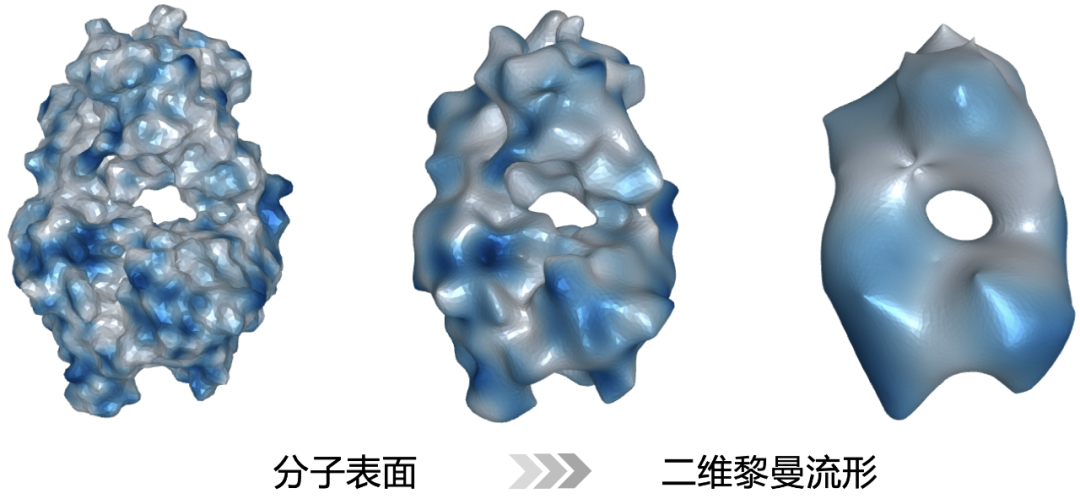
圖:抗體蛋白 (PDB ID: 6W41) 表面流形在不同分辨率下的表示。顏色代表分子表面靜電勢能函數,藍色為負電荷區域。
生物分子的表面通常是指該分子在溶液中與溶劑(例如水分子)形成的界面。我們可以將這樣的界面表示為一個三維空間中的二維黎曼流形 (Riemannian manifold)。上圖展示了一個抗體蛋白在不同分辨率下的表面流形結構,流形上的顏色對應分子表面的靜電勢能函數分布。換句話說,流形的結構勾勒了分子的形狀,而流形上分布的函數則可以表示分子表面的化學性質。因此,利用黎曼流形可以統一地表示分子的幾何結構和化學性質,從而整合分子信息并幫助 AI 模型更好地學習蛋白質的結構 - 活性關系。
在黎曼流形的表示下,每個分子都有一套獨特的「形狀基因」(Shape-DNA) 。這些形狀基因被定義為分子表面流形的拉普拉斯-貝爾特拉米算子 (Laplace-Beltrami Operator, LBO) 的本征值的集合 {???},它們提供了一種描述分子形狀的內蘊方法,不受三維空間中分子的位置和取向影響。
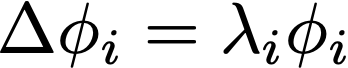
圖:黎曼流形上的拉普拉斯特征方程。Δ 代表拉普拉斯-貝爾特拉米算子,φ?和???分別代表第 i 個本征函數和對應的本征值。不同的分子表面流形有其獨特的特征方程的解。
這些形狀基因所對應的 LBO 本征函數 {φ?} 構成了分子表面流形上的一組(標準正交的)基函數 (orthonormal basis functions)。我們可以簡單將它們類比為正弦 / 余弦函數組成的傅立葉基組在黎曼流形上的拓展。因此,這些形狀基因和基函數可以幫助我們在分子表面進行調和分析 (harmonic analysis),也就是將一個流形上的函數展開成一系列基函數的線性組合的形式(下圖)。對于同一個分子表面,不同的化學性質(如疏水性、靜電勢能)可以非常簡潔地表示為一組線性組合系數(一維數組 [c?, c?, c?,...])。
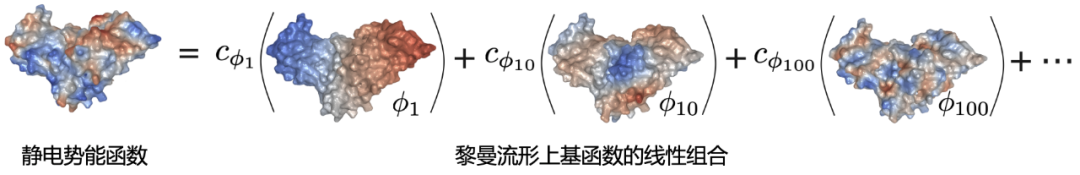
圖:分子表面黎曼流形上的調和分析。可以將流形上的函數展開成一系列基函數線性組合的形式。僅選擇性展示了 3 個基函數,其實所有的展開項都參與線性組合。
這種黎曼流形的表示是對分子表面的直接建模,得到的形狀基因和 LBO 基函數受不同離散化建模方式的影響較低(例如分子表面預處理過程中的離散采樣密度和三角剖分方式 [2]),因此增加了建模的魯棒性。
§2 分子表面幾何深度學習
上一章主要介紹了文章如何利用黎曼流形建模分子表面的幾何結構(對應拼圖的形狀),這一章我們簡要描述如何訓練神經網絡學習分子表面的化學性質(對應拼圖上的紋理)。
利用分子表面的形狀基因和 LBO 基函數來建模分子的幾何、化學性質的方法為我們提供了一種新的分子表示學習思路。基于表面的分子建模專注于對分子外部特性的描述,在一些下游任務上(比如蛋白質相互作用)可能比基于氨基酸或全原子建模的三維圖神經網絡 (Euclidean graph neural networks) 更具有優勢。「形狀基因」是對分子表面整體進行不同頻率(或粒度)信號的建模,不需要提前對表面區域進行預切割處理 [2],也讓模型適合學習不同尺度 (multi-scale) 的分子信息。
基于這套思路,團隊設計了一套幾何深度學習的方案用于分子表面上的全局信息傳遞,從而幫助神經網絡學習到更豐富的分子表面幾何、化學性質表示。文中提出了流形調和信息傳遞 (manifold harmonic message passing) 的新方法:一種類似于熱擴散的流形表面信息傳遞機制,但建模上更為靈活。熱擴散機制可以看成是對信號進行低通濾波操作,因此低頻信號比高頻信號更容易被傳播開 [3];而流形調和信息傳遞允許對不同頻率的波段進行過濾,實現高頻信號的獨立傳播 [4],因此傳播距離更遠,表示粒度更細。結合殘差連接等神經網絡結構,文中提出的方法可以學習分子表面不同尺度、不同距離的幾何與化學信息,從而對分子有更好的建模能力。
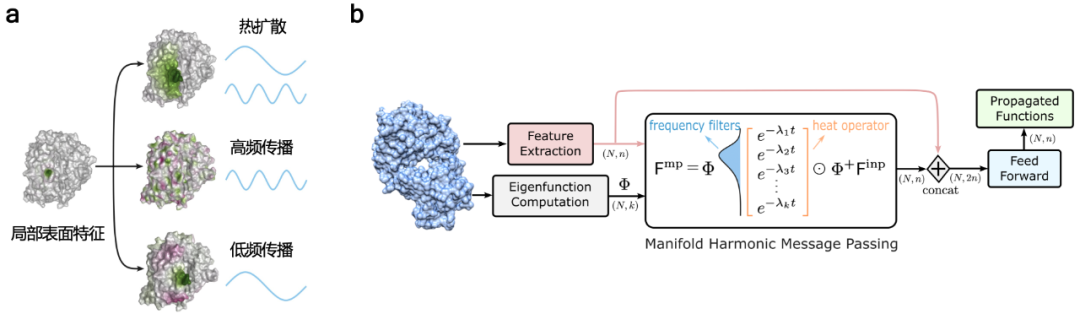
圖:利用流形調和信息傳遞機制進行分子表面的表示學習。(a)分子表面的局部特征可以通過不同的信息傳遞機制在流形上傳遞,實現有效的幾何、化學性質表示。(b)流形上的信息傳遞神經網絡結構。
§3 蛋白質拼圖 AI 模型
現在,我們有了(§1)基于黎曼流形的分子幾何表示方法和(§2)神經網絡學習到的分子表面化學性質函數這兩種工具,接下來可以利用它們來解決蛋白質復合物的構型預測問題。
具體而言,給定受體蛋白和配體蛋白的結構,我們希望預測出它們結合后復合物的構型。這里主要涉及兩個子問題:(1)結合位點在哪里;(2)受體 - 配體以什么樣的空間姿態相結合。文中提出,蛋白質之間的結合位點應該具備幾何結構互補和化學匹配這兩個重要條件,因而蛋白質復合物構型預測可以看成是一種「蛋白質拼圖」問題。類似于人類解決拼圖的思路:首先找到兩塊拼圖之間的拼接面(預測結合位點),再利用形狀和花紋的相似性旋轉其中一塊拼圖放置到正確的位置(分子對接)。
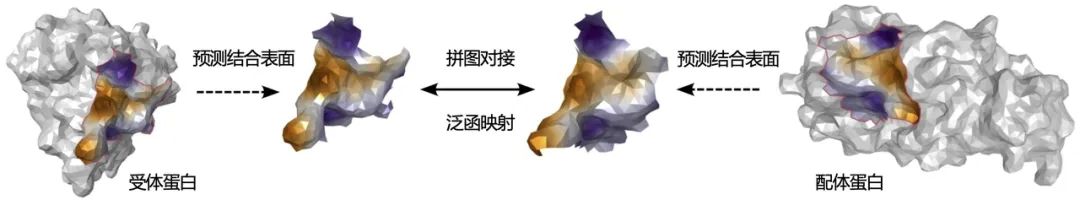
圖:「蛋白質拼圖」流程示意圖:模型首先預測兩個蛋白質之間的結合表面區域,再利用神經網絡所學函數的匹配性實現高選擇性的拼圖。
在具體的模型構建上,團隊首先利用文中提出的 HMR 模塊進行表面位點的預測 (binding site prediction)。通過流形信息傳遞機制實現分子表面的特征學習,并引入交叉注意力機制 (cross attention) 實現蛋白質分子間的信息交流。該模塊最終的輸出是模型對分子表面的某個區域是否屬于蛋白結合位點的二元預測。可以理解為這一步對應的是尋找拼圖上缺失的一塊。
接下來進入分子對接模塊:預測得到的蛋白結合表面上應具有一定的函數對應關系 (functional correspondence),因為分子間相互作用是由匹配的化學作用(如靜電力和氫鍵)決定的。也就是說能夠匹配的拼圖上所對應的紋理也應該有一致性。根據這樣的假設,團隊進一步利用泛函映射技術 (functional map) 將這種函數對應關系轉化為受體 - 配體結合位點之間的空間對應關系,并使用 Kabsch 算法將配體分子進行空間上的旋轉和平移,最終得到對接后的蛋白質復合物結構。
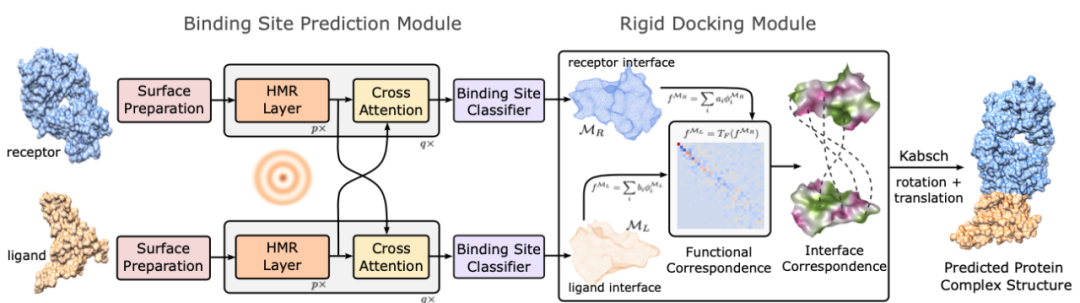
圖:「蛋白質拼圖」模型流程圖,主要分為結合表面預測(左??)和剛性分子對接(右??)兩個模塊。
文中將這種「蛋白質拼圖」方法和三維圖神經網絡的方法 EquiDock [1] 以及基于「采樣 - 篩選」策略的傳統方法(ATTRACT 和 HDOCK)進行了比較。在蛋白質對接標準測試集 Docking Benchmark 5.5 上,「蛋白質拼圖」的方法在各個衡量項目上均比三維圖神經網絡取得了更好的效果,甚至接近于一些傳統方法。而這種基于深度學習的方法預測分子對接的速度比傳統方法提高了 100 倍以上。
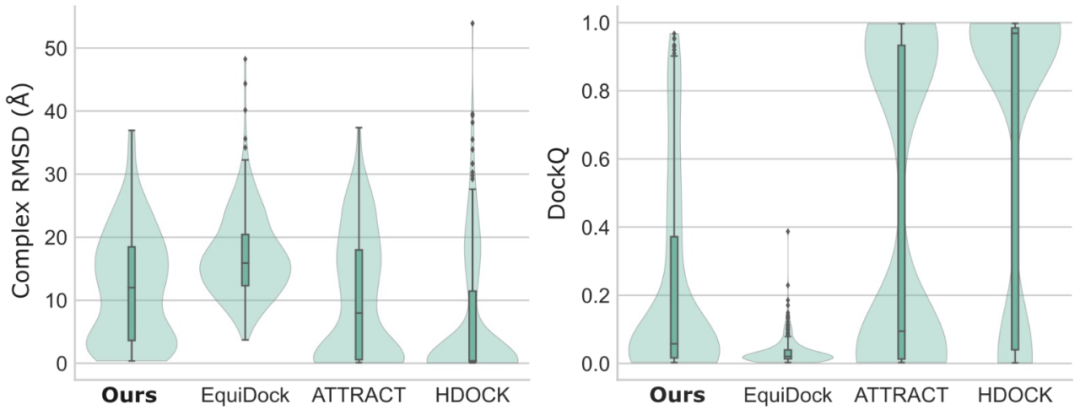
圖:「蛋白質拼圖」模型(顯示為 ours)和基于三維圖神經網絡的深度學習模型 SOTA (EquiDock) 以及傳統剛性分子對接模型 ATTRACT 和 HDOCK 的比較。Complex RMSD 越小、DockQ 越高說明預測的蛋白質復合物結構越接近真實結構。
§4 總結
這項工作提出了一種新的基于分子表面的深度學習建模方式,利用黎曼流形和形狀基因對化學、物理以及幾何相關的性質進行建模,并在蛋白質分子剛性對接的任務上超越基于三維圖神經網絡的深度學習方法。總結來說:
- - 不同于常見的基于序列或者二維 / 三維圖結構的建模方式,團隊采用了基于表面的分子建模方法。這種建模思路既保留了分子整體的三維結構,又避免了對于大分子內部結構的冗余建模。在和蛋白質功能、蛋白質相互作用等表面相關的下游任務中可能更具優勢。
- - 采用黎曼流形直接對分子表面進行建模,并用分子形狀基因和對應的本征函數來表示表面的函數分布。這種建模方法不受表面離散采樣和三角剖分方法的影響,因而具有更好的魯棒性。團隊同時展示了基于調和分析和濾波概念的表面傳播深度學習模塊,以及基于泛函映射的表面對接方法。
- - 文中利用這種表面建模方法和「拼圖」的思想實現了基于深度學習模型的蛋白質剛性分子對接。相比于目前基于三維圖神經網絡的深度學習模型取得了更好的效果,在速度上相比于傳統方法也有大幅提升。