實現10倍提升!昇思MindSpore SPONGE套件助力核磁共振蛋白質動態結構解析加速
近日,華為與高毅勤教授團隊、田長麟教授團隊、王申林教授團隊合作,基于昇騰AI基礎軟硬件平臺與昇思MindSpore AI框架開發了核磁共振波譜法(NMR)數據自動解析方法FAAST(iterative Folding Assisted peak ASsignmenT),利用昇思MindSpore SPONGE生物計算套件實現了NMR數據解析時間從數月到數小時的縮短,與現有方法相比效率提升超過10倍。
蛋白質是生命活動的主要承擔者,獲取蛋白質結構是現代藥物研發流程中不可或缺的環節。由于藥物一般在溶液態環境下與目標蛋白結合發揮作用,蛋白質的溶液態(動態)結構對于藥物研發與生命活動研究具有重要意義。
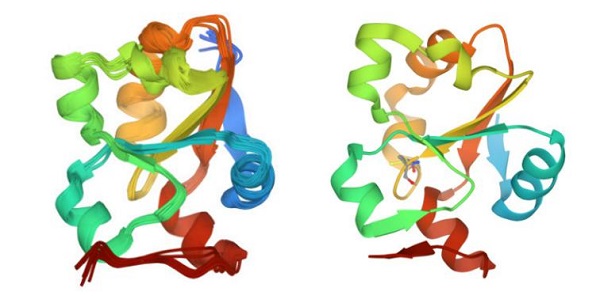
蛋白質YgaP的硫氰酸酶結構域
核磁共振方法測得的溶液態結構(左)與X射線法測得的晶體態結構(右)
已有的蛋白質結構計算方法如MEGA-Fold/AlphaFold以預測單一構象為主[1][2],在模型預測信息和實驗信息不一致時難以處理兩者誤差。如何應用AI模型輔助實驗結構解析、應用實驗信號幫助提高結構預測精度等問題依然亟待解決。核磁共振方法(NMR)是唯一一種以原子分辨率解析更貼近蛋白質在實際環境下的溶液態構象與動態結構的方法 [3],然而該方法存在數據解析速度慢的問題,平均單條蛋白需領域專家投入至少數月,而其中大部分時間都消耗在實驗數據的解析和歸屬上。
為了提高 NMR 實驗數據解析的速度和準確性,華為與高毅勤教授團隊(昌平實驗室、北京大學化學與分子工程學院和生物醫學前沿創新中心(BIOPIC))、田長麟教授團隊(中國科技大學、中科院強磁場科學中心)、王申林教授團隊(華東理工大學生物反應器工程國家重點實驗室)合作,基于昇騰AI和昇思MindSpore開發了NMR數據(NOESY譜)自動解析方法FAAST(iterative Folding Assisted peak ASsignmenT)[4],實現了NMR數據解析時間從數月到數小時的縮短,相關代碼已通過在開源社區Gitee的MindSpore SPONGE 代碼倉開源[5]。
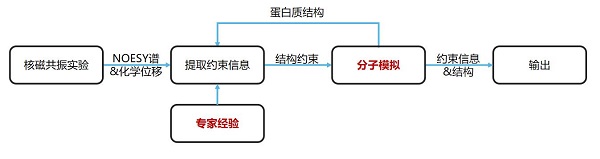
現有方法核磁共振數據解析流程
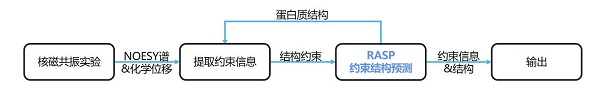
FAAST核磁共振數據解析流程
FAAST方法融合了自主創新的AI+約束結構預測模型RASP(Restraints Assisted Structure Predictor),該模型可以生成滿足NMR實驗測得的結構約束的蛋白質結構。基于RASP模型,FAAST可以實現數據解析和結構預測的交互迭代,自動解析NMR數據并獲取蛋白質動態結構與結構約束信息,在解析精度與領域專家解析持平的前提下,實現了NMR數據解析時間從數月到數小時的縮短。
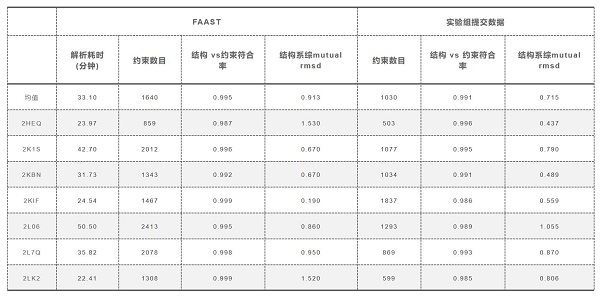
解析精度對比
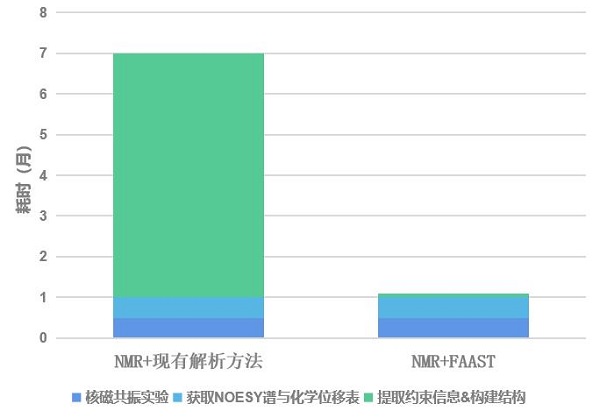
核磁共振方法各階段耗時對比
FAAST 方法大幅降低了 NMR 實驗數據解析的門檻。應用該方法,領域專家可以從繁瑣的數據解析工作中釋放精力,專注于實驗設計和所得結構的生物學分析,推動生命科學研究和藥物研發流程取得新突破。
北京大學博雅特聘教授、北大-清華生命科學聯合中心研究員唐淳教授認為,該工作不僅可以將核磁共振實驗約束引入到結構預測,還可以用來引入其他的約束。如質譜交聯[6]、熒光共振能量轉移[7]都可以作為距離約束,可以全面互補的來對蛋白結構優化。因此,高毅勤團隊所開發的這一方法也會在整合結構生物學得到廣泛的應用。
[1]Jumper J, et al. Highly accurate protein structure prediction with AlphaFold. Nature. 2021 Aug;596(7873):583-589. doi: 10.1038/s41586-021-03819-2. Epub 2021 Jul 15. PMID: 34265844; PMCID: PMC8371605.
[2]Liu S, Zhang J, Chu H, et al. PSP: million-level protein sequence dataset for protein structure prediction[J]. arXiv preprint arXiv:2206.12240, 2022.
[3]Lutomski CA, El-Baba TJ, Robinson CV, Riek R, Scheres SHW, Yan N, AlQuraishi M, Gan L. The next decade of protein structure. Cell. 2022 Jul 21;185(15):2617-2620. doi: 10.1016/j.cell.2022.06.011. PMID: 35868264.
[4Liu S, Chu H, Xie Y, et al. Assisting and Accelerating NMR Assignment with Restrainted Structure Prediction[J]. bioRxiv, 2023: 2023.04. 14.536890.
[5]https://gitee.com/mindspore/mindscience/tree/master/MindSPONGE/applications/research/FAAST
[6]Stahl, K., et al., Protein structure prediction with in-cell photo-crosslinking mass spectrometry and deep learning. Nat Biotechnol, 2023.
[7]Tang, C. and Z. Gong, Integrating Non-NMR Distance Restraints to Augment NMR Depiction of Protein Structure and Dynamics. J Mol Biol, 2020. 432(9): p. 2913-2929.