單模型斬獲「蛋白質突變預測」榜一!西湖大學提出基于結構詞表方法 | ICLR 2024 Spotlight
蛋白質結構相比于序列往往被認為更加具有信息量,因為其直接決定了蛋白質的功能。
而隨著AlphaFold2帶來的巨大突破,大量的預測結構被發布出來供人研究使用,如何利用這些蛋白質結構來訓練強大且通用的表征模型是一個值得研究的方向。
西湖大學的研究人員利用Foldseek來處理蛋白質結構,將其編碼成一維的離散token,并與傳統的氨基酸進行結合,形成了結構感知詞表(Structure-aware Vocabulary),以此將結構信息嵌入到模型輸入中,增強模型的表征能力。

論文地址:https://www.biorxiv.org/content/10.1101/2023.10.01.560349v4
Github地址:https://github.com/westlake-repl/SaProt
在預訓練上,本文使用了目前最多的蛋白質結構(大約4000萬),在64張A100上訓練了3個月,最終開源了具備650M參數量的模型SaProt(同時包括了35M的版本)。實驗結果表明SaProt各種蛋白質任務上都要好于之前的序列和結構模型。
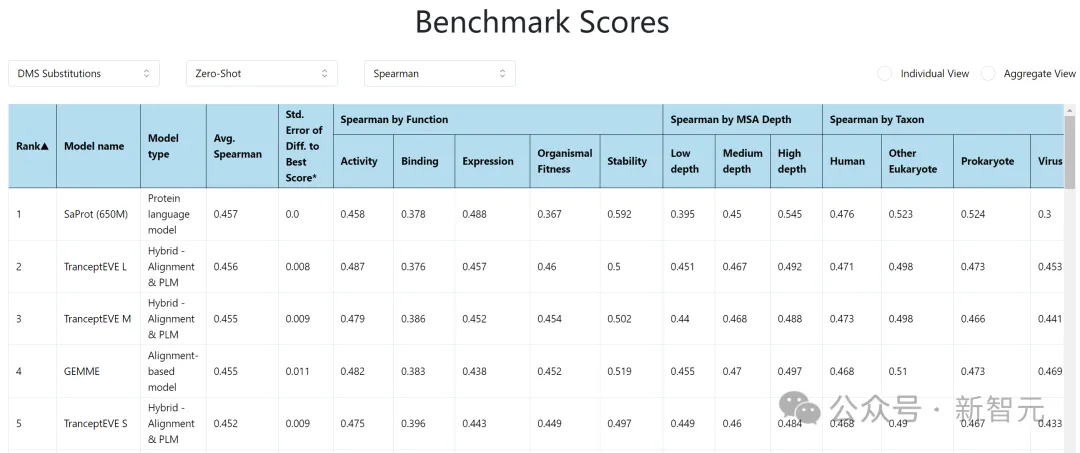 ProteinGym:https://proteingym.org/benchmarks
ProteinGym:https://proteingym.org/benchmarks
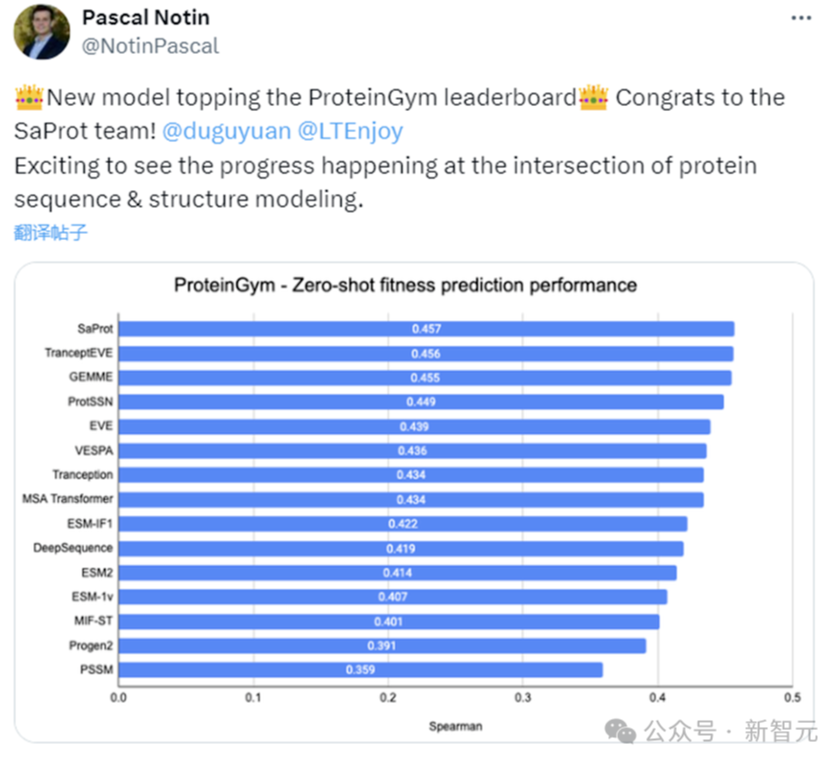
方法
該研究利用Foldseek將蛋白質進行編碼,生成了一維的3Di結構序列(使用了Foldseek的結構詞表,每種3Di token代表不同的局部結構),這樣的結構序列與氨基酸序列是等長的。
因此研究人員使用了一種簡單而有效的結構嵌入方式:將結構詞表和氨基酸詞表計算笛卡爾積(即兩兩組合),形成新的結構感知詞表。
這樣對于蛋白質的每個位點,其氨基酸類型和對應的局部結構都能組合成新詞表中的某個元素,從而讓模型同時考慮到蛋白質的序列與結構信息。
本文使用Bert架構進行掩碼語言建模(Masked Language Modeling )預訓練(關于訓練的更多細節請參考原論文)。

結構感知詞表
實驗
方法對比
一個可能令人疑惑的問題就是為什么需要這樣編碼結構?論文展示了使用不同的結構編碼方式進行預訓練的結果圖:
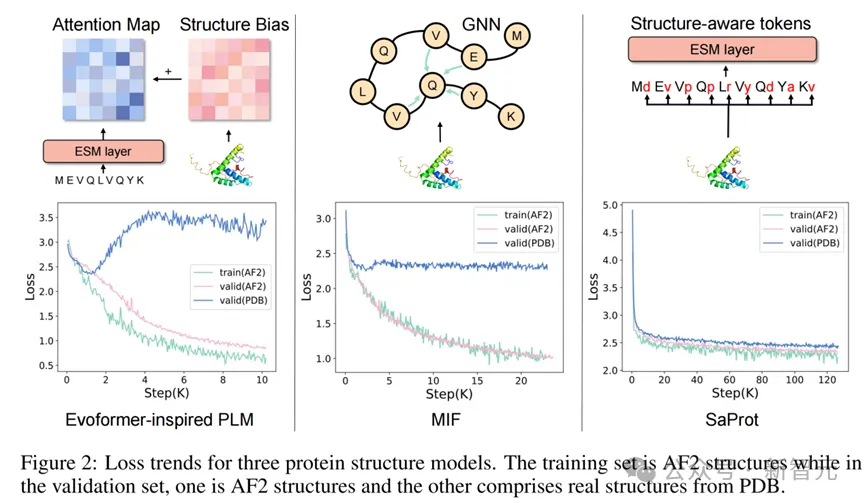
不同結構模型訓練的loss曲線圖
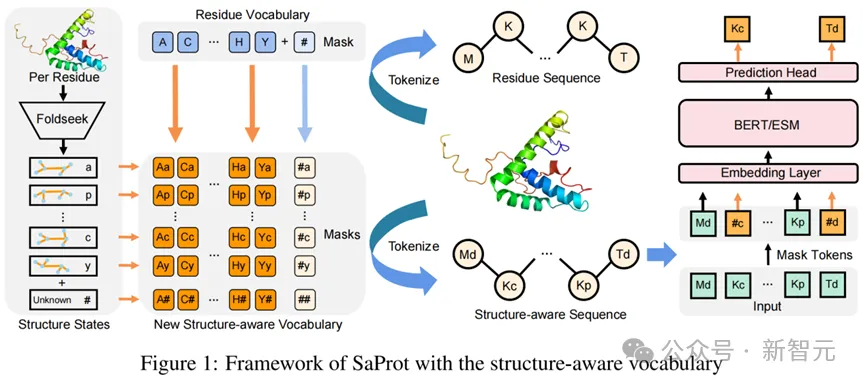
圖左和圖中是兩種經典的蛋白質結構建模方式,即將結構信息編碼成bias后添加到transformer的attention map中(如Evoformer,Uni-Mol),或者使用圖神經網絡的方式建模蛋白質的空間關系(如MIF,GearNet等)。
然而從loss圖中可以發現,當上述兩種建模方式在AF2結構上使用MLM的訓練目標進行預訓練時,模型會非常迅速地過擬合(表現為在AF2預測結構上預測loss非常低,但在PDB真實結構上loss停滯甚至上升)。
作者推測這是由于AF2預測出來的蛋白質結構帶有一些隱藏的模式(patterns),由于前兩種方式是直接對蛋白質的三維坐標進行建模,這些隱藏的pattern可能很輕易地就被模型識別出來,從而造成了信息泄露的問題,讓模型無需真正學習到蛋白質的進化信息就能輕松地完成訓練目標。
而結構感知詞表通過將蛋白質結構編碼成一維的結構序列,在盡可能保留結構模式的情況下忽略了精細的坐標數值,因此模型能夠有效地利用結構信息而不受到隱藏pattern的影響。
Zero-shot測試
作者在蛋白質突變數據集(ProteinGym)上和真實人類臨床疾病數據集(ClinVar)上測試了SaProt的zero-shot能力,結果如下:
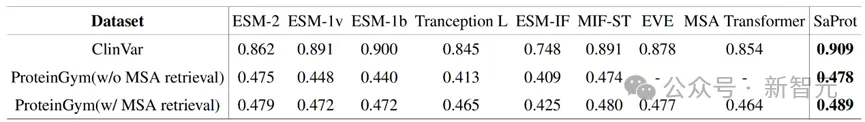
Zero-shot實驗結果
SaProt在兩個數據集上都超越了以往的所有結構和序列模型,證明了其在zero-shot預測突變上具備優異的能力。
監督微調測試
本文還涵蓋了各種下游任務來測試模型表現,結果如下:
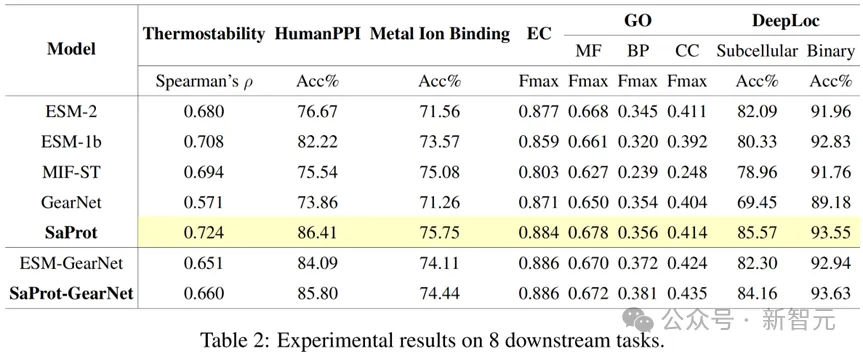
下游任務fine-tune結果
SaProt在各個下游任務上都超越了以往的序列和結構模型,展示出了其強大且通用的表征能力。
結構信息測試
SaProt在4000萬的蛋白質結構上進行訓練,獲得了強大的表征能力。一個可能的疑問是如何確定SaProt學到了更多的結構信息而不是模型被訓練得更好?論文對SaProt和ESM-2在殘基接觸預測任務(Contact Prediction Task)上進行了測試。作者凍住了模型的backbone,只訓練一個線性分類層。
實驗結果如下:
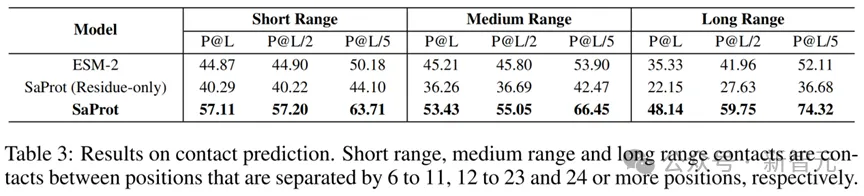
Contact Prediction Task的結果
從結果可以看到,由于結構token的嵌入,SaProt的表現大大超越了ESM-2,這表明SaProt蘊含了非常豐富的結構信息,使其能夠在結構預測任務上獲得十分優異的結果。
同時,論文在SCOPe數據庫上對alpha蛋白質和beta蛋白質進行了可視化,結果如下:
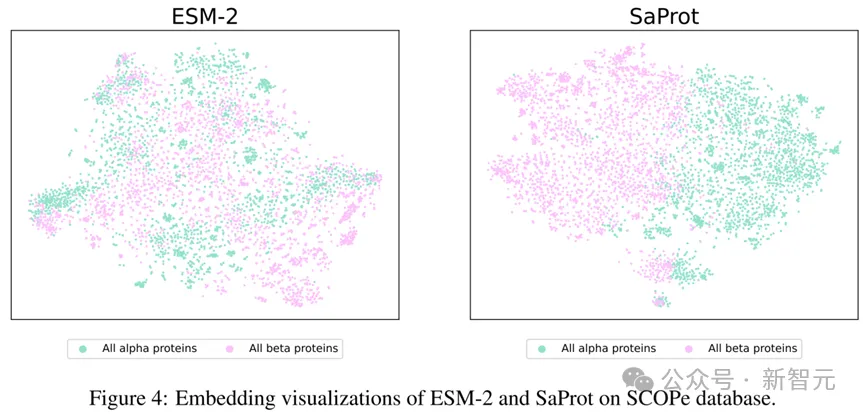
在SCOPe數據庫上的Embedding可視化
SaProt的可視化結果非常清晰地將alpha蛋白質和beta蛋白質區分開來,而ESM-2的可視化結果卻將兩種蛋白質混雜在一起,這說明了SaProt對結構的變化有很強的感知能力。
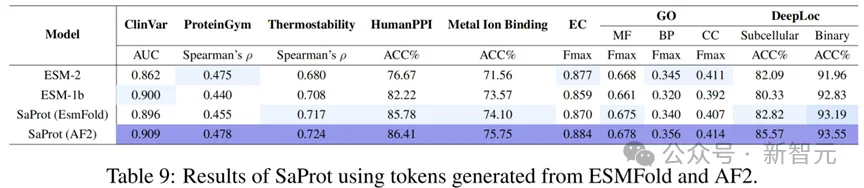
不同結構預測方法的比較
除了AF2,目前還存在許多其他的單序列結構預測方法(如ESMFold),因此本文額外測試了其他方法預測出來的結構對SaProt性能的作用。
結果如下:
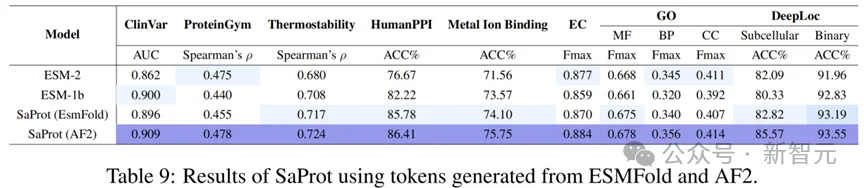
不同結構預測方法的fine-tune結果
從測試結果可以看出,雖然SaProt在AF2結構上的表現非常好(模型本身也是基于AF2結構進行訓練的),但其他的結構預測方法也能讓SaProt與ESM-2等模型性能相當。
這意味著考慮到計算與時間成本,單序列結構預測模型也能作為替代方法輸入到SaProt中。
局限
雖然SaProt經過訓練展示出了優異的性能,但依然還有一些可以改進的地方,例如:
1. Foldseek默認的結構詞表大小只有20,如果有更加精準的結構編碼模型,擴大結構表征的詞表大小,是不是能進一步提升模型利用結構的能力?
2. 由于計算能力的限制,SaProt只在650M上完成了訓練。如果能夠繼續擴大模型規模,是否可以進一步地提升模型表現?
3. 論文雖然已經測試了很多的蛋白質任務,但還有一些其他任務可以應用探索,例如蛋白質序列設計(給定backbone預測氨基酸序列)等。








































