AlphaFold2原理:注意力機制取代卷積網絡,預測準確性提升超30%
本文經AI新媒體量子位(公眾號ID:QbitAI)授權轉載,轉載請聯系出處。
最近,DeepMind開源AlphaFold2,讓學術圈再一次沸騰了。
這意味著,對于普通研究人員而言曾需要花幾年時間才能破解的蛋白質結構,現在用AlphaFold2幾小時就能算出來了!
那么,如此厲害的AlphaFold2究竟如何做到的呢?
DeepMind團隊已經將它的詳細信息在《Nature》上公開發表。

現在,就讓我們來看看AlphaFold2的魔法是怎么實現的吧。
卷積消失了,Attention來了
論文中,研究人員強調AlphaFold2是一個完全不同于AlphaFold的新模型。
的確,它們使用的模型框架都不一樣,這也是AlphaFold2準確性能夠突飛猛進的主要原因。
此前AlphaFold中所有的卷積神經網絡,現在都被替換成了Attention。
為什么要這樣做呢?
我們首先要了解一下AlphaFold的工作原理:
它主要是通過預測蛋白質中每對氨基酸之間的距離分布,以及連接它們的化學鍵之間的角度,然后將所有氨基酸對的測量結果匯總成2D的距離直方圖。
然后讓卷積神經網絡對這些圖片進行學習,從而構建出蛋白質的3D結構。
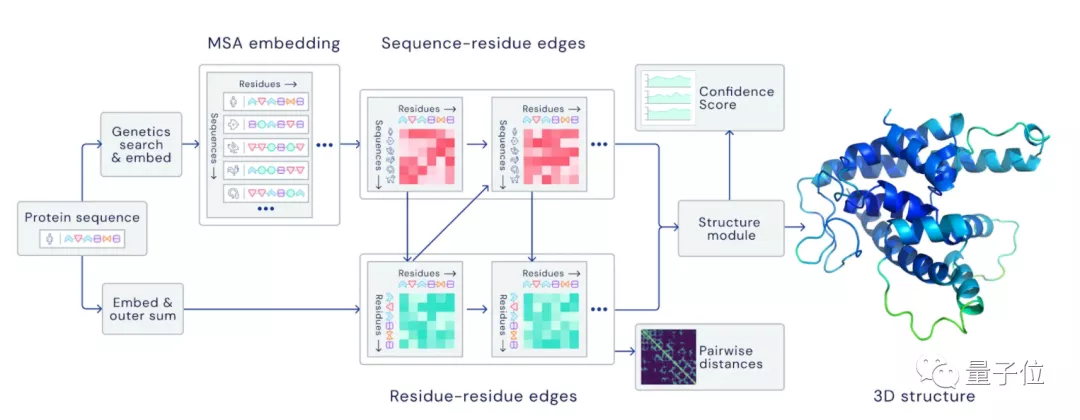
△AlphaFold主要架構
但這是一種從局部開始進行預測的方式,很有可能會忽略蛋白質結構信息的長距離依賴性。
而Attention的特點剛好可以彌補這一缺陷,它是一種模仿人類注意力的網絡架構,可以同時聚焦多個細節部分。
這樣可以使得框架預測的結果更加全面、準確。
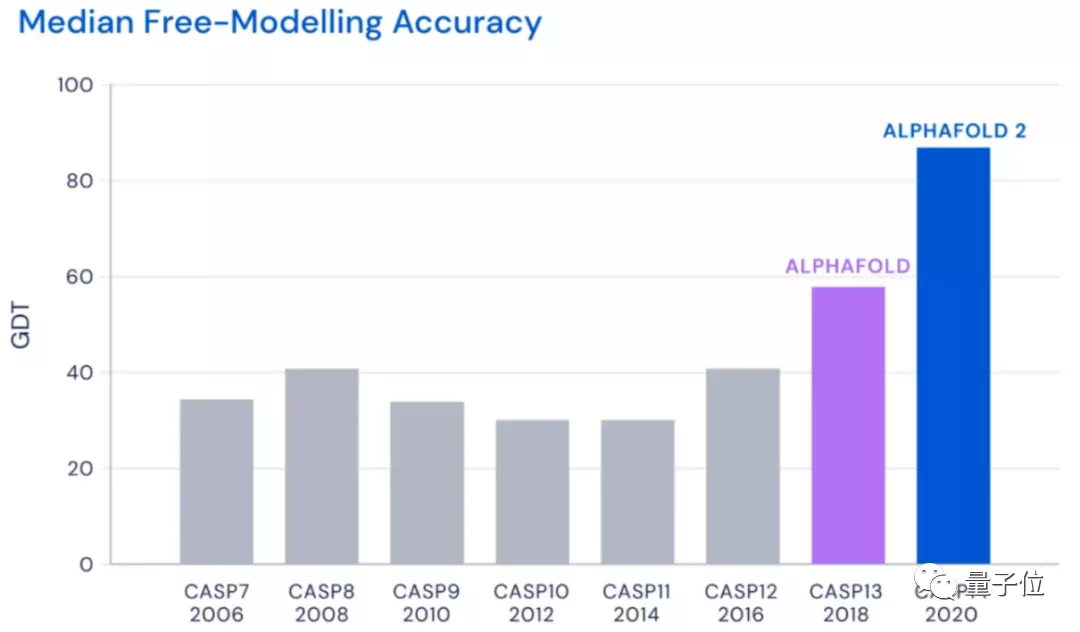
在CASP13中,AlphaFold預測的準確性還只有不到60分。
但是在CASP14中AlphaFold2就將準確性直接拔高到了92.4/100。
圖網絡+Attention
具體來看,AlphaFold2主要利用多序列比對(MSA),把蛋白質的結構和生物信息整合到了深度學習算法中。
它主要包括兩個部分:神經網絡EvoFormer和結構模塊(Structure module)。
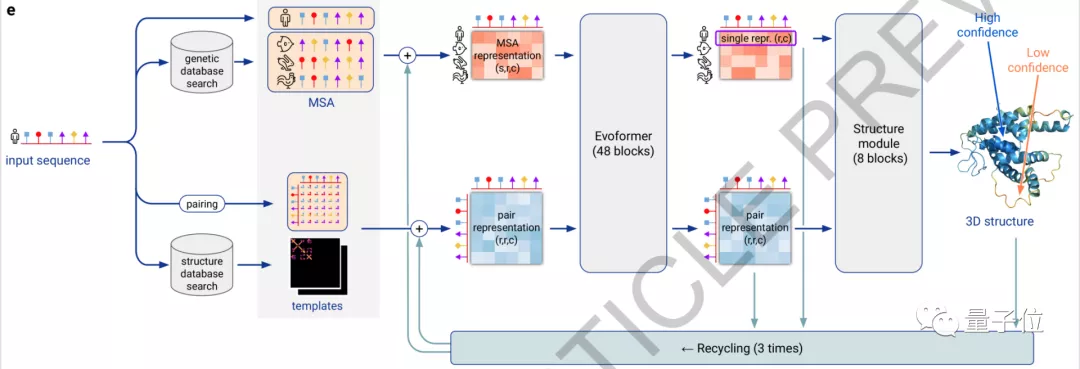
在EvoFormer中,主要是將圖網絡 (Graph networks)和多序列比對 (MSA)結合完成結構預測。
圖網絡可以很好表示事物之間的相關性,在這里,它可以將蛋白質的相關信息構建出一個圖表,以此表示不同氨基酸之間的距離。
研究人員用Attention機制構建出一個特殊的“三重自注意力機制(Triangular self-attention)”,來處理計算氨基酸之間的關系圖。
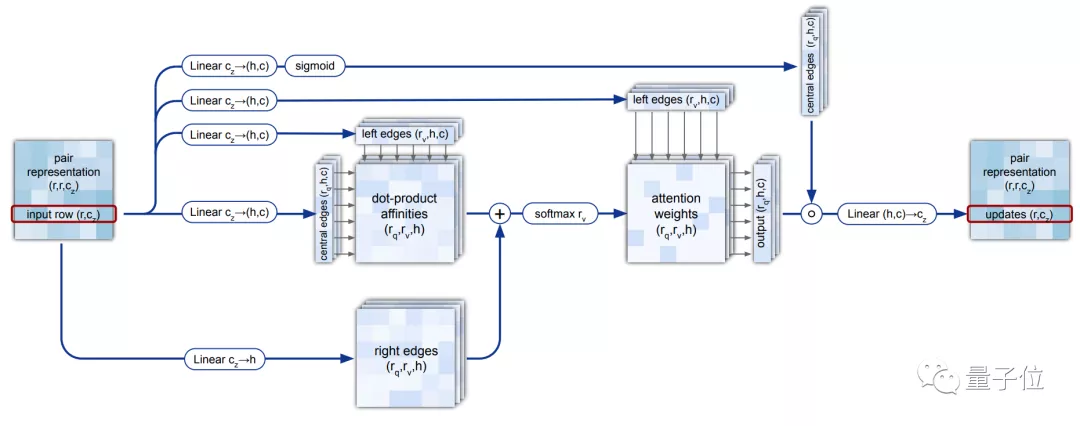
△三重自注意力機制(Triangular self-attention)
然后,他們將這一步得到的信息與多序列比對結合。
多序列比對主要是使相同殘基的位點位于同一列,暴露出不同序列之間的相似部分,從而推斷出不同蛋白質在結構和功能上的相似關系。
計算出的氨基酸關系與MSA進行信息交換,能直接推理出空間和進化關系的配對表征。
預測所有原子的3D結構
架構的第二部分是一個結構模塊 (Structure Module),它的主要工作是將EvoFormer得到的信息轉換為蛋白質的3D結構。
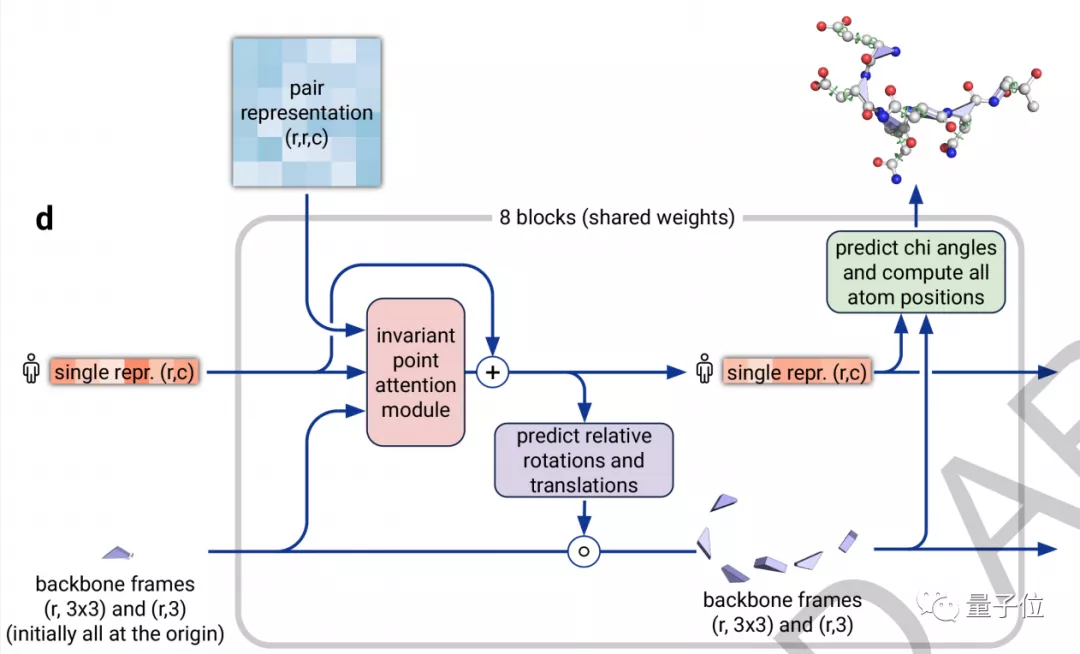
△結構模塊(Structure module)
在這里,研究人員同樣使用了Attention機制,它可以單獨計算蛋白質的各個部分,稱為“不變點注意力(invariant point attention)”機制。
它以某個原子為原點,構建出一個3D參考場,根據預測信息進行旋轉和平移,得到一個結構框架。
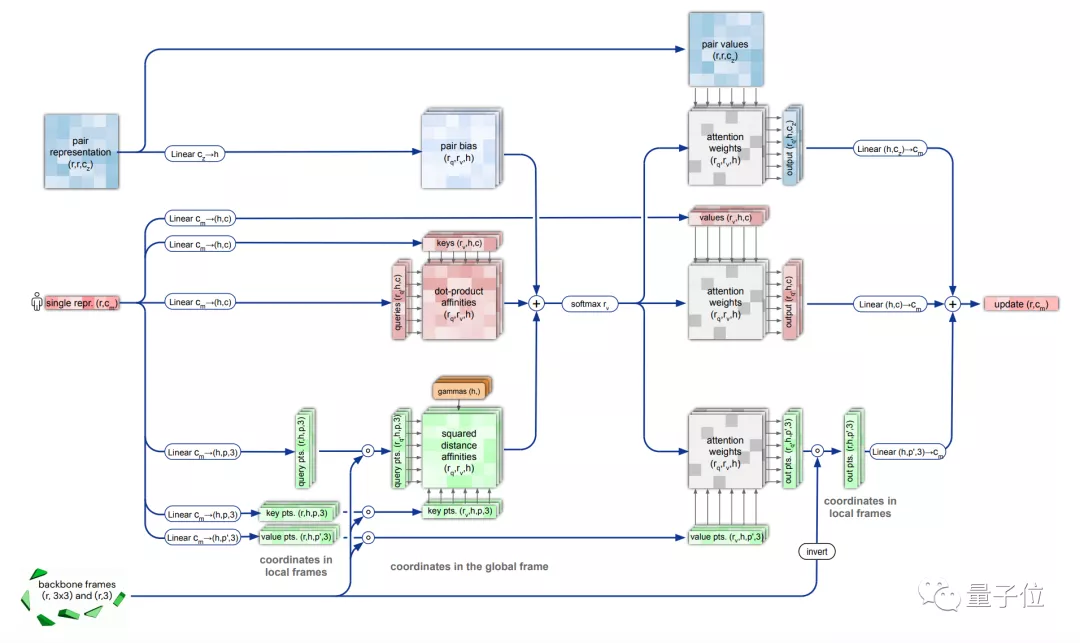
△不變點注意力(invariant point attention)
然后Attention機制會對所有原子都進行預測,最終匯總得出一個高度準確的蛋白質結構。
此外,研究人員還強調AlphaFold2是一個“端到端”的神經網絡。
他們會反復把最終損失應用于輸出結果,然后再對輸出結果進行遞歸,不斷逼近正確結果。
這樣做既能減少額外的訓練,還能大幅提高預測結構的準確性。
為破解蛋白質折疊謎題帶來希望
Alphafold2的出現,能更好地預判蛋白質與分子結合的概率,從而極大地加速新藥研發的效率。
此次Alphafold2開源,將進一步推動科學界前進。
據了解,目前DeepMind已經與瑞士的一些研究團隊合作,通過預測蛋白質結構開展藥物方面的研究。
事實上,研究Alphafold2預測程序本身,也為探索蛋白質結構折疊原理帶來了希望。
芝加哥大學的計算生物學家Jinbo Xu就表示:
這些工具的開源,意味著科學界能夠在此基礎上開發出更加強大的軟件。
論文地址:
https://www.nature.com/articles/s41586-021-03819-2_reference.pdf
補充材料:
https://static-content.springer.com/esm/art%3A10.1038%2Fs41586-021-03819-2/MediaObjects/41586_2021_3819_MOESM1_ESM.pdf*








































