使用深度學習進行醫療影像分析:文件格式篇
今年 3 月,英偉達的 GTC 2017 大會上展示了很多深度學習技術在醫療領域中的卓越工作。Ian GoodFellow、Jeremy Howard 以及其他的深度學習專家都分享了他們對深度學習的見解。頂尖的醫科學校(例如西奈山醫院、紐約大學、麻省綜合醫院等)以及肺癌 BOWL 的獲獎者 Kaggle 一起解釋了他們的建模策略。回顧我們的系列文章,在上一篇文章中,我們討論了在文本和圖像數據上的基礎深度學習。在本文中,我們會聚焦于分析醫療圖像和它們的格式。
這篇文章由三部分組成–醫療圖像和組成、醫療圖像格式,以及醫療圖像格式的轉換。本文的目標就是組織知識來幫助我們實現最終目標–用深度學習進行醫療圖形的分析。
醫療圖像和組成
與這個主題相關的一個很好的資源來自意大利生物組織和生物圖像研究所(IBB)的 Michele Larobina 和 Loredana Murino 發表的論文。這個研究所是意大利國家研究委員會(NCR)的一部分。它是意大利最大的公共研究機構。另一個可供參考的資源是這篇論文:「Working with the DICOM and NIfTI Data Standards in R」
那么,醫療圖像是什么呢?醫療圖像是對解剖區域的內部結構和功能的一種表征。它以二維像素或者三維體素的形式呈現出來。映射到空間位置的數值是對采樣過程和重建過程的離散表征。用來描述一個確定采樣模態視野的像素數量是對解剖結構和功能的細節的表達。像素表達的數值取決于成像模式、采樣協議、重建以及后續處理過程。
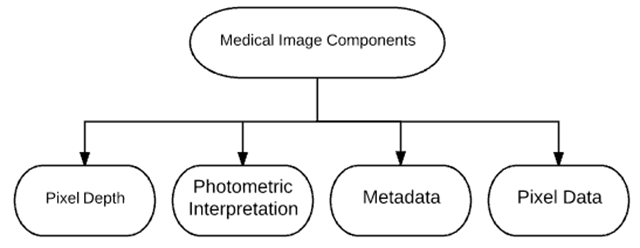
醫療數據的組成
醫療數據的組成
如圖所示:醫療數據有四個關鍵的組成部分–像素深度、光度解釋、元數據以及像素數據。這幾部分決定了圖像的大小和分辨率。
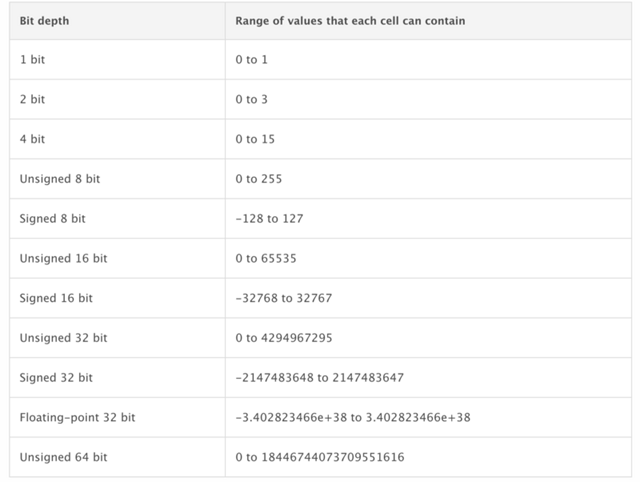
像素深度(Pixel Depth)或者位深度(Bit Depth)或者色深度(Color Depth)就是用來編碼每一像素的信息所用的位數。例如,一個 8 位的柵格會擁有從 0 到 255 這 256 種各不相同的數值。
參考:http://desktop.arcgis.com/en/arcmap/10.3/manage-data/raster-and-images/bit-depth-capacity-for-raster-dataset-cells.htm
光度解釋具體化了像素數據被解釋成正確的圖片展示的方式,如單色圖像或者彩色圖像。為了確定像素值中是否存儲了彩色信息,我們引入了每個像素的樣本的概念,也就是大家都知道的通道數量。單色圖像每個像素只有一個樣本,圖片中并沒有存儲彩色信息。我們使用從黑色到白色的灰度級別來展示這種圖片。灰度的數量明顯取決于用來存儲這個樣本的位數,在這種情況下,與像素深度是一致的。像 X 光片、CT 和磁共振這樣的放射醫療影像都有一個灰度光度解釋。核醫學圖像都以彩色的形式展現,例如 PET 和 SPECT。
元數據就是圖片中所描述的信息。它看上去可能是很奇怪的,但是無論在什么格式的文件中,都存在一些超越像素數據并且和圖像相關的信息。這類被稱作元數據的信息通常都以頭部的形式儲在文件的起始部分,它至少會包含以下信息:圖像矩陣的維度、空間精度、像素深度以及光度解釋。
像素數據–這里存儲的是像素數值的大小。根據不同的數據類型,像素數據可能以整型或者浮點型的類型存儲,使用表達數據所需的最少的數據位。
所以,圖像的大小=頭部大小(包含元數據)+行×列×像素深度×幀的數量
醫療圖像的格式
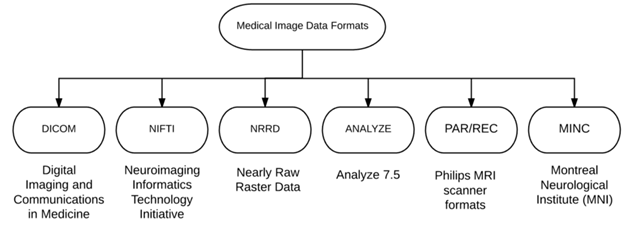
放射生物圖像中主要有六種格式–DICOM(醫療中的數字圖像和通信),NIFTI(神經影像學信息技術計劃),PAR/REC(飛利浦 MRI 掃描格式),ANALYZE(Mayo 醫療成像)以及 NRRD(近乎原始光柵數據)和 MNIC 格式。
截止 2017 年 5 月的醫療圖像格式
其中 DICOM 和 NIFTI 是最常用的格式。
DICOM 格式的基本知識
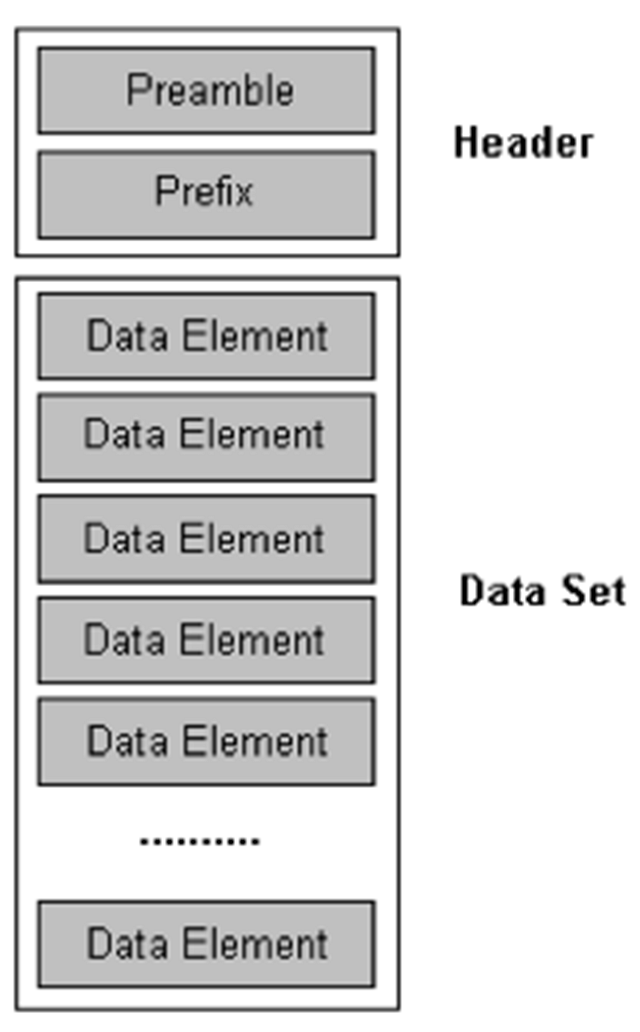
DICOM 代表的是醫療數字成像和通信。DICOM 是由美國國家電氣制造商協會(NEMA)制定的標準。它定義了醫療成像領域中關于信息處理、存儲、打印以及傳輸的標準。這些都是你在掃描儀或者某家醫院的圖片歸檔和通信系統(PACS)能夠立即得到的文件格式。
它包括了文件格式和能夠接收圖像和 DICOM 格式的病人數據的實體之間使用 TCP/IP 進行通信的協議。
一個 DICOM 文件包含文件頭部和同文件名的*.dcm 圖像數據。文件頭部的大小取決于它所提供的信息的多少。文件頭包含以下信息:病人的 ID,病人的姓名,圖像的模態以及其他信息。它定義了幀的數量以及圖像的精度。這些信息會被圖像瀏覽器在顯示圖像時用到。對于一個單詞采樣,會有很多個 DICOM 文件。
來源:https://www.leadtools.com/sdk/medical/dicom-spec1
pydicom 是一個讀取 dicom 文件的 python 庫。可以參考本文第一部分的示例代碼。
用深度學習的方法進行醫療圖像分析
深度學習可以分析圖像和視頻,并將分析結果用于很多種應用中,例如無人駕駛……
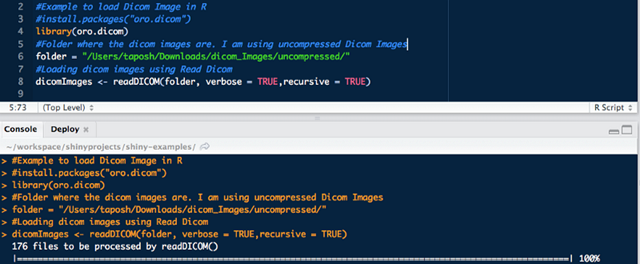
oro.dicom 是一個可以讀取 dicom 數據的 R 語言包。
上圖所示是用 oro.dicom 包來讀取一個已經解壓了的 DICOM 文件。
NIFTI 格式基本知識
Nifti 格式最初是為神經影像學發明的。神經影像信息學技術計劃(NIFTI)將 NIfTI 格式預設為 ANALYZE7.5 格式的替代品。它最初的應用領域是神經影像,但是也被用在其他領域。這種格式的主要特點就是它包含兩個能夠將每個體素的索引(i,j,k)和它的空間位置(x,y,z)關聯起來的仿射坐標。
nibabel 是一個能夠讀取 nifti 文件的 Python 庫,oro.nifti 是一個能夠讀取 nifti 文件的 R 語言庫。
DICOM 和 NIFTI 的區別
DICOM 和 NIfTI 這兩種格式的主要區別是:NIfTI 中的圖像原始數據被存儲成了 3 維圖像,而 dicom 一些 2 維的圖層。這就使得 NIFTI 更加適合那些應用在 DICOM 上的機器學習的方法,因為它是以 3D 圖像建模的。處理一個單獨的 NIFTI 文件要比處理成百上千個 dicom 文件更加容易一些。與 DICOM 格式下的好多個文件相比,NIFTI 格式下,每個 3d 圖像只有兩個文件。
Nrrd 格式的基本知識
靈活的 Nrrd 格式包含一個單獨的頭文件和若干個可以被結合在一起或者分開的圖像文件。Nrrd 頭文件為科學可視化和圖像處理準確地表征了 N 維光柵信息。國家醫療圖像計算聯盟(NA-MIC)開發了一種使用 NRRD 格式來表征擴散加權圖像(DWI)體積和擴散張量圖像(DTI)的方法。為了目視確認張量的方向和期望的神經解剖學的結構一致,Nrrd DWI 和 Nrrd DTI 數據能夠被讀取為 3D 切片。
通用的 NRRD 文件格式(包括數據和文件頭):
參考: http://teem.sourceforge.net/nrrd/format.html#general.1
MINC 格式的基本知識
MINS 代表的是醫療圖像 NetCDF 工具包。
Minc2 從 NetCDF 變換成了 HDF5。HDF5 支持無限制多的數據類型,并且它就是為靈活高效的 I/O 和大容量的復雜數據而設計的。這些附加的特征有助于 Minc2 處理大量的復雜數據集。
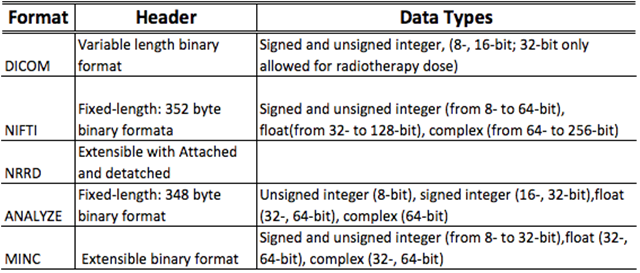
從研究論文中得到的一些關于這幾個格式的對比:
參考:Medical Image Formats, Springer Publication 2014. 醫療圖像格式
格式轉換
dicom 轉換成 NIFTI
dicom2nii(https://www.nitrc.org/projects/dcm2nii/)是一個用來把 DICOM 轉換為 NIFTI 的工具。nibabel 是一個讀寫 nifiti 文件的 python 庫。如果你你想把 DICOM 轉換成 NIFTI,可以使用自動轉換的工具(例如,dcm2nii)。python2 下的庫 dcmstack 可以將一系列的 DICOM 文件堆疊成多維數組。這些數組能夠被寫成 NIFTI 的文件,同時還加上一個可選的頭部擴展,這個頭文件包含原始 DICOM 文件的所有元數據。python3 提供了一個新的用來完成這個格式轉換的庫–dicom2nifti。我建議讀者去查看一下 nipy 這個項目。
DICOM 到 MINC 的轉換
腦成像中心(BIC)的 MINC 團隊開發了將 DICOM 轉換為 MINC 格式的工具。這個工具是用 C 語言開發的,它在 GitHub 上的地址為:https://github.com/BIC-MNI/minc-tools/tree/master/conversion/dcm2mnc
NIfTI 或 ANALYZE 轉換成 MINC
腦成像中心(BIC)的 MINC 團隊還開發了將 NIfTI 或 ANALYZE 轉換成 MINC 格式圖像的工具。這個程序叫做 nii2mnc。包括 nii2mnc 的很多格式轉換工具在這里可以看到:https://github.com/BIC-MNI/minc-tools/tree/master/conversion。
總結
我們可以看到,目前已存在幾種用于存儲醫療圖像并且可以用在深度學習中的文件格式。我們的目標是使用最佳的文件格式,該格式能夠讓我們得到卷積神經網絡(CNN)所需的所有特征,以實現準確的預測。