ICLR 2024 | UIUC等提出BioBridge:用知識圖譜構建多模態生物醫學基礎模型
01 引言
這是 UIUC Zifeng Wang 等發表在 ICLR'24 上的論文。

論文題目:
BioBridge: Bridging Biomedical Foundation Models via Knowledge Graphs
論文鏈接:
??https://arxiv.org/pdf/2310.03320.pdf??
在生物醫學研究領域,一直以來,基礎模型(Foundation Models,簡稱FMs)大多局限于單一模態的數據處理,比如僅僅專注于蛋白質序列、小分子結構或者是臨床數據分析。這種單模態的處理框架雖然在特定任務上表現出色,但在應對多樣化的生物醫學數據時,其潛能卻受到了限制。
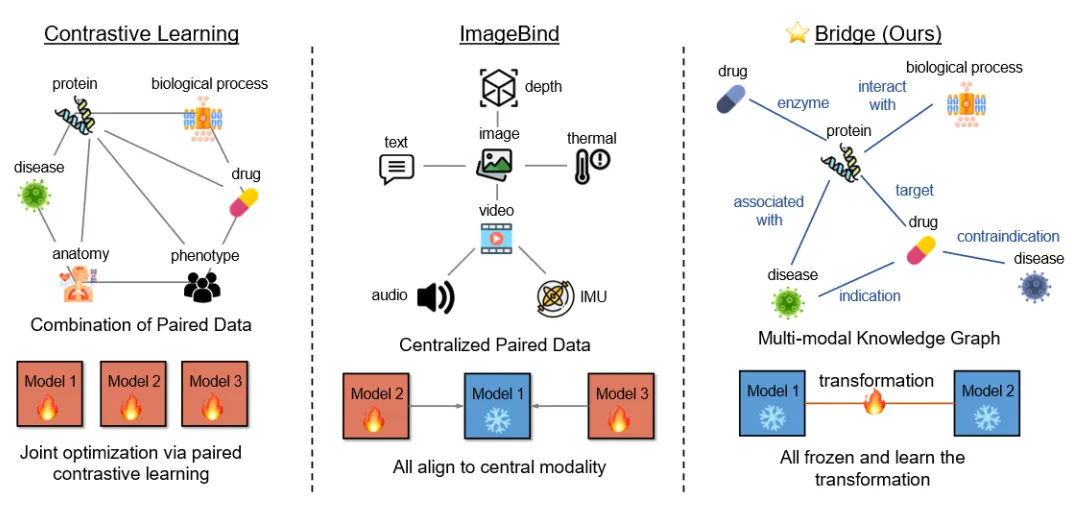
▲ 幾種構建多模態模型的方法:CLIP,ImageBind,BioBridge
02 CLIP
傳統的多模態方法,比如大名鼎鼎的 CLIP 模型 [1],大多建立在配對的多模態數據上。比如,如果我們要訓練一個蛋白質-文本模型,就需要收集大量的蛋白質序列和其對應的文本描述,從而做對比學習。這一類工作有很多,比如在 x-rays 和臨床筆記上做對比的 MedCLIP [2],在文本和蛋白質上做的 ProteinDT [3],在文本和分子上做的 MolT5 [4],等等。
但是,假如我們有超過兩個模態,比如有三個模態 A,B,C,在這種思路下,我們就需要三種配對數據,AB, AC, 和 BC,才能訓練一個三塔的多模態模型。當存在更多模態的時候,這種對于數據的需要是排列組合式增長的。這就使這種配對數據的思路很難用于超過兩個模態的情況。
03 ImageBind
ImageBind [5] 是 Meta AI 提出的一種用來構建多模態檢索模型的一種方法。其本質是選擇一個模態作為中間模態,比如圖片,然后讓其他模態的模型都向圖片模態靠攏。在這情況下,假設我們有一個中間模態 A,還有另外兩個模態 B,C,我們就需要收集 AB 和 AC 兩種配對數據。在這種情況下,ImageBind 一共需要 (模態數-1)個數的配對數據,而且需要更新 (模態數-1)個模態的編碼器來構建多模態系統。
04 BioBridge
本文提出用知識圖譜(KG)來作為多模態數據源,從而將獨立訓練的單模態基礎模型橋接(Bridge)成一個能夠處理多種數據模態的系統(Multimodal FM)。
單模態的數據往往比多模態數據的數量要大的多。我們可以收集到超過 2.5 億個蛋白質序列或者 15 億個分子結構,但是只能收集到 44 萬個蛋白質文本對。大模型的縮放原理告訴我們,基礎模型需要“大”數據才能產生所謂的 emergent ability。所以我們更容易訓練出很好的單模態模型,卻不容易從零開始訓練出一個多模態模性。
在這種思路下,我們希望能夠在訓練好的單模態基礎模型的基礎上,去把它們合并在一起去作為一個多模態的系統。這就產生了這篇文章里提到的 “bridge” 的概念。
KG 存在許多三元組,比如(蛋白質 A, associated with,疾病 B)。這樣,我們希望能夠用一個單模態的蛋白質模型去編碼蛋白質 A,然后設計一個關系模型取編碼 “associated with”,將蛋白質 A 的embedding 映射到疾病的 space 里。這樣,我們可以匹配由蛋白質 A 生成的 embedding 和疾病 B 的 embedding,做到跨模態檢索。
通過 BioBridge,不同類型的生物醫學數據能夠實現無縫交互和轉換,而且這一切都不需要對原有的單模態模型做任何調整。
05 一些結果
這里放一小部分結果,更多的結果在原文可見。
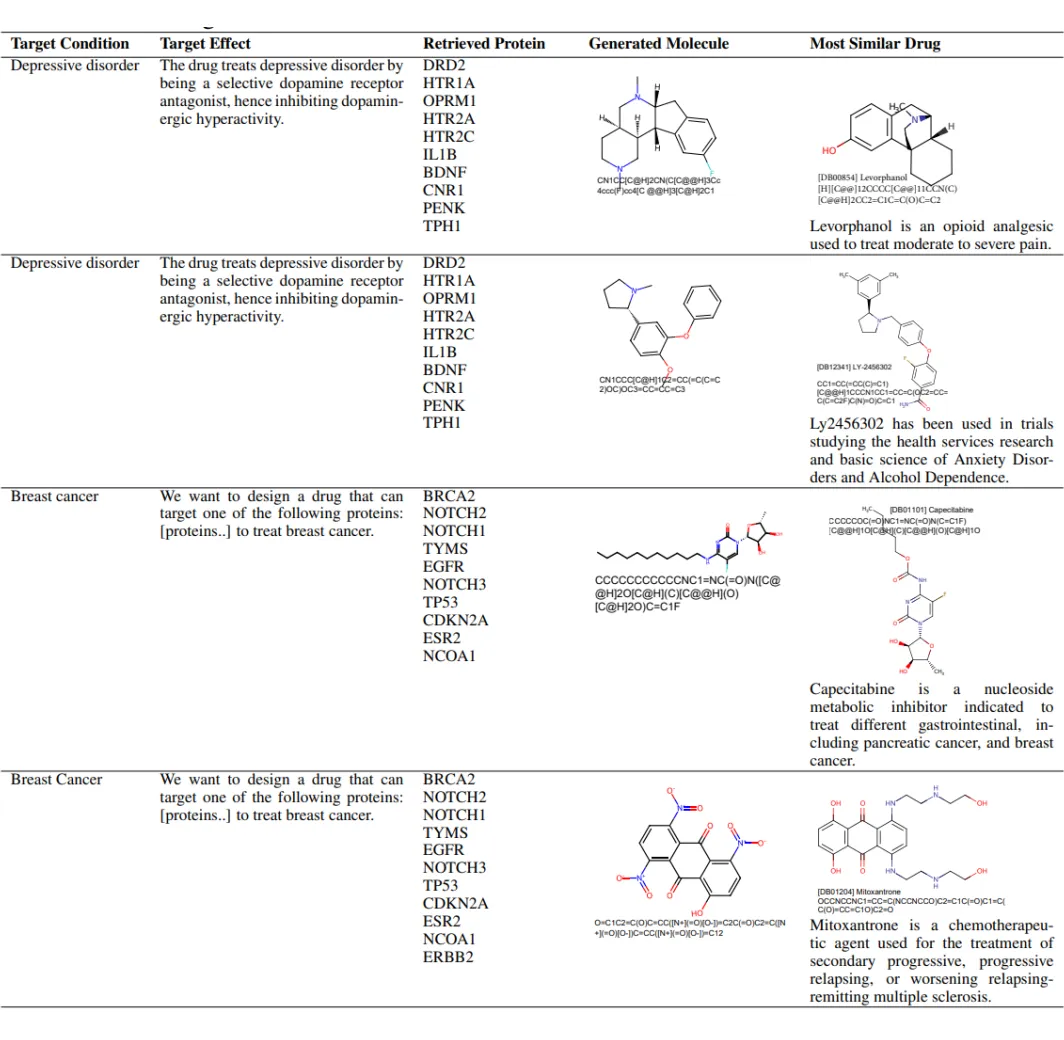
這里我們是用 BioBridge 來根據輸入的文本,檢索相關的蛋白質,再用蛋白質+文本去 prompt Galactica 生成分子模型。這里的思路是利用 BioBridge 來構建一個多模態的 RAG 系統。本文發現加上 RAG 的 galactica 能夠更好地生成符合輸入文本描述的分子。
本文轉自 PaperWeekly ,作者:Zifeng Wang

















